De fleste mennesker har svært ved at forstå, at universet har 100 milliarder stjerner. Det faktum, at menneskekroppen huser 400 gange så mange mikrober, er derfor næsten ufatteligt. At forstå, hvordan de fungerer og interagerer, kræver derfor meget specielle værktøjer. I processen omkring udforskningen af nye biologiske territorier har professor Peer Bork udviklet en perlerække af computerbaserede værktøjer, der kan analysere de mindste biologiske enheder. De har gjort det muligt for ham selv og forskere og virksomheder over hele verden at udvikle behandlinger og bæredygtige produktionsmetoder. For dette banebrydende arbejde modtager han Novozymes-prisen 2021.
De fleste mennesker forbinder ikke hype med nøjagtig videnskab. Ikke desto mindre er videnskab ofte baseret på hype – populære og interessante teorier, der desværre ikke altid består den ultimative videnskabelige test: de eksperimentelle beviser. Peer Bork har i stedet for at prøve at bevise teorier dedikeret sin videnskabelige karriere til at afkode de mange eksperimentelle data, der i dag i stigende grad oversvømmer videnskabens verden. Det har krævet konceptuelt helt nye tilgange.
”Hype er meget almindeligt indenfor biologi, og nogle forskere analyserer data med en bestemt hypotese i tankerne. Jeg stræber konstant efter at udforske disse nye felter og skabe de værktøjer, der gør det muligt for data at vise os, hvad der faktisk foregår. Dette har gjort det muligt for mig at skabe en måde til bedre at forstå, hvordan mikroorganismer interagerer i vores kroppe, så vi kan diagnosticere og helbrede sygdomme, og i verdenshavene, hvor dette hjælper os med at forstå og vedligeholde biodiversitet,” forklarer Peer Bork, direktør for Heidelberg-filialen af European Molecular Biology Laboratory (EMBL) i Tyskland.
Det lette liv i det vestlige Tyskland
Selvom Peer Bork har tilbragt det meste af sin 34-årige karriere i Heidelberg, startede hans karriere faktisk et andet sted: i det tidligere Østtyskland – i Leipzig – og senere i Berlin, hvor hans ph.d.-vejleder Jens Reich var en af de førende figurer i den østtyske modstand mod det kommunistiske styre. At forske skabte derfor undertiden uventede udfordringer.
”Det var lige før, Berlinmuren faldt. Jens Reich var under konstant overvågning af Stasi, og det var meget sandsynligt, at han ville ende i fængsel. Det var derfor risikabelt at tage ham som vejleder. Og hvis jeg ville læse en ny videnskabelig artikel, tog det ofte uger at få den, fordi biblioteket havde meget få tidsskrifter. Vi var derfor nødt til at tigge forfatterne om genoptryk, hvilket gjorde tingene vanskelige og langsommelige. Så min tid som forsker lærte mig ikke at tage noget for givet,” erindrer Peer Bork.
Tiden med Jens Reich lærte også Peer Bork om vigtigheden af at have en engageret og støttende vejleder og altid behandle alle lige. Men lige så vigtigt lærte hans tid som forsker i Østtyskland ham også, at det at være talentfuld er en ting, men at det i sidste ende er det engagement, man udviser, der giver gode resultater.
”Livet var så let i det vestlige Tyskland sammenlignet med det hårde liv i Østtyskland dengang – eller for den sags skyld i lavindkomstlande i dag. Så når jeg ansætter ph.d.-studerende, vælger jeg ikke bare de bedste kandidater. Jeg vil kunne føle deres drive og energi – at de virkelig vil opnå noget. Så jeg tror bestemt, at den manglende sikkerhed i Østtysland og det at skulle arbejde hårdt for at få succes, påvirkede mig stærkt. Engagementet var afgørende. Ellers kunne du simpelthen ikke opnå noget,” fortæller Peer Bork.
Svarende til LEGO
Peer Borks eget engagement blev skabt gennem interessen for matematik og datalogi i begyndelsen af 1980'erne, da han første gang blev udsat for computerprogrammering på en gammel russisk computer på størrelse med en bil.
"Computeren kunne ikke engang dividere to numre korrekt og havde brug for at blive fodret af udstansede bånd," husker han.
Da han senere kom til at studere ved universitetet i Leipzig, blev Peer Bork fascineret af potentialet til at løse biologiske problemer beregningsmæssigt. Det begyndte med at simulere og optimere enzymproduktionsprocesserne med differentialligningssystemer. Senere var det enzymerne og deres funktionalitet, der fascinerede ham.
”Aktiviteten af enzymerne viste sig at afhænge af kofaktorer – små molekyler, der aktiverer enzymerne. Derefter blev jeg fascineret af den evolutionære udvikling af de områder eller domæner i enzymerne, hvor de små molekyler binder, da dette er nødvendigt for at forstå optimering af effektiv enzymfunktion," siger Peer Bork.
Det viste sig, at enzymerne af interesse alle var modulære, hvilket betyder, at de kofaktorbindende domæner var ens på tværs af meget forskellige enzymer, men dette var alt sammen stort set ikke undersøgt på det tidspunkt.
"Så jeg begyndte at samle sekvenser af de her enzymer, fokuserede på domænerne, der fungerede som en LEGO-blok i en mere kompleks struktur, og forsøgte så at finde lignende domæner i nye sekvensdatabaser," tilføjer Peer Bork.
Da eksisterende software ikke var tilstrækkelig til dette, programmerede Peer Bork og en kollega et nyt værktøj (PAT) baseret på sekvensmønstrene. Med det kunne han lave sine første virkelige biologiske opdagelser, der viser den udbredte karakter af disse bindende domæner. Sekvensanalysefeltet var dog stadig ret lille i slutningen af 1980'erne og begyndelsen af 1990'erne, men databaserne blev hurtigt udvidet.
Da Peer Bork fandt biologi fascinerende og havde en naturlig flair for computere og sekvenssammenligninger, var det sjovt og relativt nemt for ham at komme på en anden biologisk innovation.
”Jeg vendte snart fra bare at undersøge de specifikke forudsigelser af bindingssteder i enzymdomæner til at identificere mange andre typer domæner i andre typer proteiner, da det viste sig, at dette LEGO-princip om modularitet var meget udbredt, især i ekstracellulære proteiner. Så jeg begyndte at samle sekvensmønstre – "signaturer" af disse. Vi begyndte derfor at opbygge en database med det formål at udforske proteiners domæneopbygning, da vi nu vidste, at hvert domæne har en bestemt underfunktion," fortæller han.
Det første smarte værktøj
Som mange andre fra det gamle Østtyskland flyttede Peer Bork vestpå, da muren faldt. I 1990 fik han med med økonomisk støtte først en besøgende og snart efter en fast stilling ved European Molecular Biology Laboratory (EMBL) i Heidelberg, en universitetsby gennemsyret af det sydvestlige Tysklands traditioner og et flagskib inden for europæisk molekylærbiologisk forskning med mere end 100 uafhængige forskningsgrupper på seks steder.
"Ånden og åbenheden var overvældende, når man lige var ankommet fra Østtyskland. Atmosfæren var ikke kun multikulturel. Man samarbejdede også. Du kunne altid bede en af dine kolleger på en anden etage om at hjælpe dig. Noget, der normalt ville tage mig 5 år at teste selv, tog pludselig kun et par uger på EMBL. Og fordi du kun har lov til at blive der i maksimalt 9 år, er der altid friske nye ansigter og inspiration, hvilket er fantastisk. En kontinuerlig strøm af talentfulde unge mennesker med helt nye ideer," husker Peer Bork.
Peer Bork fremstod hurtigt som en talentfuld ung forsker, og hans evne til at kombinere computere med biologi gav pote. I midten af 1990'erne udvidede han og hans kolleger en elektronisk samling af ekstracellulære domæner, han havde med sig fra Berlin. De designede nu et simpelt, men yderst effektivt værktøj, SMART (Simple Modular Architecture Research Tool), til at forudsige proteindomæners funktioner effektivt via de sekvensdatabaser, der var i hastig vækst.
"Værktøjet var også rigtig nyttigt i de tidlige dage i sekventering af hele genomer, hvor de fleste proteiner ikke havde nogen funktion forbundet med dem. Her var SMART en stor hjælp til at forudsige det ved at sammenligne sekvenser fra mange klassificerede domæner af signalproteiner. På den måde kunne vi kvantificere deres forekomst i hele genomer. For eksempel opdagede vi, at næsten 7 % af generne i gærgenomet indeholdt et eller flere signaldomæner - 350 mere end tidligere estimeret. Og da World Wide Web netop var opstået, byggede vi også en webressource op omkring dette, som alle kunne bruge på en intuitiv måde," siger Peer Bork.
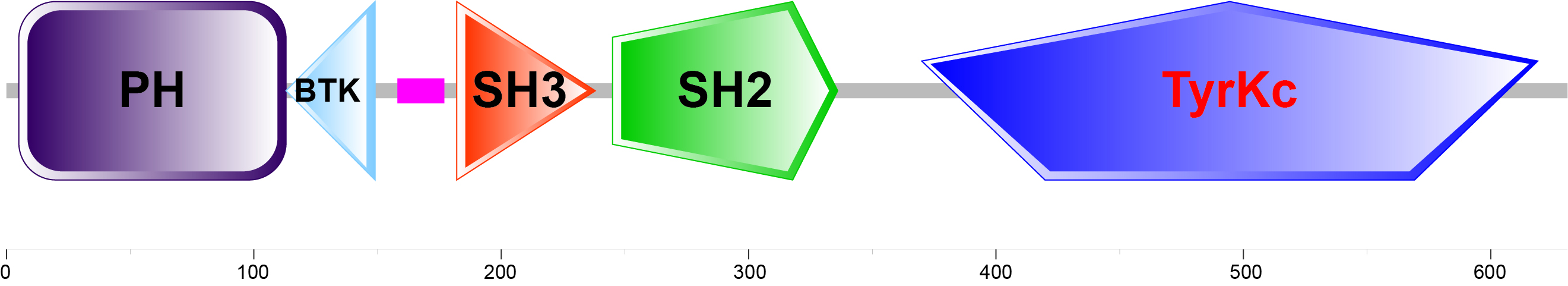
Med værktøjet SMART (simpelt modulært arkitekturforskningsværktøj) bliver hurtig identifikation og markering af domæner i proteiner mulig, så man bliver i stand til hurtigt at bestemme de modulære arkitekturer for alle genprodukter i sekventerede genomer.
Afgørende for bioteknologi
SMART var bare den første af Peer Borks mange onlineværktøjer og var et vendepunkt for ham, da det var en billet til at blive involveret i et stort gennembrud, ikke kun for ham selv, men også for hele genetikken. På grund af sit voksende ry med skabelsen af SMART blev han inviteret til at være en del af det mest banebrydende videnskabelige projekt i de sidste 50 år: det humane genomprojekt.
"Jeg er altid blevet drevet til at arbejde inden for nye forskningsområder og til at hjælpe med at udvikle de nødvendige værktøjer. Jeg kan godt lide at deltage i samarbejdende tværfaglige projekter, fordi dette udvider min horisont og skaber masser af læring, og selvfølgelig fordi de kan føre til virkelig store biologiske opdagelser. Det humane genomprojekt var et godt eksempel på det, og SMART blev et vigtigt redskab, der hjalp med at finde funktionen for mange proteiner, som det humane genom koder for," siger Peer Bork.
Erfaringerne fra analysen af det menneskelige genom gjorde samtidig, at Peer Bork blev inviteret til at hjælpe med til de fleste af de andre tidlige genomsekventeringsprojekter. Mens generne i et genom næsten kan være tilfældigt arrangeret i fx dyr, er genernes rækkefølge i bakterier stærkere begrænset på grund af bakterielle operoner og de grupper af gener, der udtrykkes sammen for at kunne udføre en funktion sammen.
”Den gentagne tilstedeværelse af gener i umiddelbar nærhed i genomerne på tværs af bakterier og arkæale arter indikerede en funktionel interaktion mellem proteinerne, de koder for. Så snart et par bakterielle genomer blev offentligt tilgængelige, oprettede vi søgeværktøjet STRING. Med en konceptuelt helt nye tilgang opdagede vi helt ny funktionalitet i stor skala: nemlig hvilken interaktionspartner et protein kodet af et bestemt gen ville have, hvilket førte til netværk af interagerende gener i et genom," forklarer Peer Bork.
Igen blev værktøjet kombineret med det voksende antal af allerede analyserede genomer, som var gjort tilgængelige på et websted ved hjælp af netværksvisualiseringer, hvilket var banebrydende på det tidspunkt. Sidenhen blev der tiføjet andre metoder til at forudsige interagerende gener og forskellige andre databaser med information om interagerende proteiner, så STRING snart indeholdt organismer fra hele livets træ, inklusive mennesker. Ved hjælp af STRING kunne forskere over hele verden nu undersøge deres egne genomiske data og udlede proteiners funktion og deres indbyrdes afhængighed.
"Da antallet af sekvenser fra forskellige organismer steg, kunne forskere og bioteknologiske virksomheder frit analysere deres egne data og lære om funktionaliteten af deres målorganismer eller individuelle proteiner. Hvis de for eksempel ønskede at få gær til at producere et bestemt humant protein, kunne de hurtigt sikre, at ikke kun det enkelte gen blev klonet, men også dem, der koder for tæt interagerende proteiner," siger Peer Bork.
Et interaktivt livets træ
Peer Bork og hans kolleger indså gennem deres analyse af bevarelsen af gensekvenser på tværs af det voksende antal fuldstændigt sekventerede organismer, at kun meget få sekvenser var fuldstændig konserverede. Det skyldes bl.a. at meget få gener faktisk er til stede i alle organismer, fra bakterier til dyr. De identificerede således kun 40 sekvenser, der dels er til stede i (næsten) alle organismer, og kun i én kopi. Kun hvis begge dele er opfyldt, tyder det på, at de udfører den samme funktion.
"Med de her 40 markør-gener som udgangspunkt kunne vi nu konstruere et livets træ på en konsistent og næsten automatisk måde – og med stor nøjagtighed. Men vi havde brug for en måde at vise forskellige træk ved træet, så ideen om et andet værktøj blev født: iTOL, det interaktive livets træ, til visualisering af grundlæggende egenskaber ved forskellige arter, fletning af forskellige typer data, tilpasning af skærmen på forskellige måder, tilføjelse af forskellige datasæt, og deling af resultater med andre," forklarer Peer Bork.
I dag har det interaktive livets træværktøj iTOL og den tilknyttede webressource mere end 20.000 individuelle brugere om måneden og gemmer mere end 1,3 mio. individuelle træer, der er lavet af brugerne for at undersøge forholdet mellem gener eller organismer fra alle Jordens riger.
"Nu, da vi havde overblikket over fuldstændigt sekventerede genomer på tværs af livets træ, og hvordan generne i disse interagerer, var den næste udfordring , om man kunne udvide visualiseringen til organismeinteraktioner, som i virkelige økosystemer, der kunne vise den enorme og næsten ukendte biodiversitet på vores planet. I stedet for blot at stole på de sekventerede individuelle organismer begyndte vi derfor den overvældende opgave at fange dem i deres naturlige sammenhæng ved at sekventere prøver af hele organismesamfund," fortæller Peer Bork.
Resultaterne er metagenomer, som er milliarder af små sekvensfragmenter fra et stort antal organismer, hvoraf de fleste var mikrobielle og ukendte. Så der var behov for masser af bioinformatik og nye værktøjer til at organisere disse puslespil i gener og organismer. En endnu større udfordring var at udvikle koncepter og metoder til at sammenligne metagenomer.
"Selvom mikroorganismer findes overalt, vidste vi meget lidt om de fleste omgivende mikroorganismer, fordi det er vanskeligt at dyrke dem under standard laboratorieforhold, og fordi de var blandet sammen, så det var vanskeligt at skelne mellem, hvilke sekvenser der stammer fra hvilke organismer, og hvor mange der kom fra hver enkelt. Med de nye metoder og analysekoncepter, der blev udviklet, kunne vi sammenligne forskellige levesteder som hav eller jord og vise, at sammensætningen af generne giver et molekylært fingeraftryk i et miljø," siger Peer Bork.

Peer Bork er direktør for Heidelberg-filialen af European Molecular Biology Laboratory (EMBL) i Tyskland
Tarmmikrobiom opdelt i tre
Evnen til pålideligt at kvantificere fordelingen af mikrober i miljøprøver kickstartede udviklingen et helt andet sted: menneskekroppen. Peer Bork oprettede sammen med europæiske kolleger Metagenomics of the Human Intestinal Tract project (MetaHIT). De indsamlede og sekventerede oprindeligt fæcesprøver fra 124 mennesker fra Danmark og Spanien – nogle raske, nogle med fedme og nogle med inflammatoriske tarmsygdomme – for at bestemme, om deres tarmmikrober var forskellige.
På det tidspunkt var folk kun lige begyndt at tale om mikrobiomet, og vi var overbeviste om, at forståelsen af, hvordan tarmmikrober påvirker menneskers sundhed, krævede at kunne sammenligne deres genetiske fingeraftryk. Hvis man på en pålidelig måde skulle kunne skelne mellem sundhed og sygdom, krævede det endnu en gang udvikling af nye beregningsværktøjer.
Disse var grundlaget for adskillige banebrydende opdagelser på det menneskelige tarmmikrobiom af METAHIT-konsortiet. Efter den første grundlæggende karakterisering af et humant tarmmikrobiom fra en større gruppe mennesker i 2010 offentliggjorde Peer Bork og hans kolleger en endnu mere bemærkelsesværdig artikel i Nature året efter og afslørede, at vores tarmsystem kan kategoriseres i tre enterotyper: Bacteroides, Prevotella og Ruminococcus. Historien blev dækket af tusinder af aviser verden over.
”Selvom hver af os sandsynligvis har mere end 1.000 bakteriearter i vores mikrobiom, kan de alle, ligesom blodgrupper, grundlæggende opdeles i disse tre enterotyper, hver med forskellige undertyper af bakterier. Selvom vi ikke ved, hvad der knytter enterotyperne sammen, ser de ud til at være relativt stabile over tid: hvis du har en særlig enterotype i dag, har du det sandsynligvis stadig et år senere,” forklarer Peer Bork.
Hvordan medicin påvirker mikrobiomet
At studere det menneskelige tarmmikrobiom åbnede mange muligheder og praktiske anvendelser. Selvom hvad der er "normalt" stadig den dag i dag er uklart, begyndte Peer Bork og hans kolleger at finde mikrobielle markører for visse sygdomme, såsom i tyktarms- eller bugspytkirtelkræft, og de viste sig at være bedre end eksisterende kræftmarkører med implikationer for tidlig sygdomsdiagnose.
”Efter vi havde beskrevet den mikrobielle signatur for tyktarmskræft, testede vi, om signalet var robust nok – også i andre kliniske og geografiske omgivelser. Vi undersøgte fækale genomer hos næsten 400 raske mennesker og 400 mennesker med tyktarmskræft og fandt et stabilt signal, der kunne være et supplement til de eksisterende ikke-invasive fækale blodprøver. Så ved at kombinere de to tilgange har vi nu forbedret testsikkerheden, så vejen er banet for en ikke-invasiv og billig tidlig screeningstest,” fortæller Peer Bork.
Forskerne har allerede patenteret tilgangen og de mikrobielle tyktarmskræftmarkører og udviklet en test. Lige nu er pilotforsøg i fuld gang sammen med et stort diagnostikfirma. De håber, at virksomheden snart vil kunne udrulle en forbedret test for tyktarmskræft. Et lignende projekt er også i gang med at udvikle ikke-invasiv tidlig diagnose af kræft i bugspytkirtlen.
I processen fandt Peer Bork og hans kolleger også ud af, at terapeutiske lægemidler i høj grad påvirker tarmmikrobiomet, ofte mere end selve sygdommen. Faktisk påvirker mere end en fjerdedel af alle lægemidler rettet mod den menneskelige krop også vores tarmmikrobiom.
”Igen var det nødvendigt at udvikle nye værktøjer for at kunne adskille virkningen af stoffer og sygdomseffekter. Vi fandt overraskende, at forskellige typer medicin påvirker den enkeltes mikrobiom på meget forskellige måder, så selv to personer med sammenlignelige profiler i deres mikrobielle artssammensætning har sandsynligvis unikke mikrobielle profiler, der reagerer forskelligt på den samme medicin,” siger Peer Bork.
Planetens mikrobiom?
Peer Bork og hans kolleger studerer i øjeblikket –også ved hjælp af deres værktøjet STITCH – de kemiske interaktioner, der er mellem lægemidler og proteiner i tarmen, og hvordan denne viden kan bruges til at optimere lægemiddelbehandling for hver person individuelt på baggrund af undersøgelse af den enkeltes tarmmikrobiom. Så igen forsøger Peer Bork altså at bygge bro mellem den grundlæggende videnskabelige opdagelse og praksis. En brobygning, der bekræftes af, at han er medstifter af hele fem bioteknologiske virksomheder.
Med mere end 650 videnskabelige artikler, 260.000 citater og et h-indeks på 214 er han utvetydigt en førende forsker inden for biovidenskab. Ikke desto mindre er han langt fra færdig med at gøre store opdagelser. I stedet fortsætter Peer Bork og hans team med at springe ind i helt nye felter og har derfor altid brug for at udvikle værktøjer, som han derefter deler, også til gavn for forskere inden for bioteknologi.
”Vores mål med mikrobiomforskning har været at forbedre folks sundhed. Som bioinformatiker bliver komplekse prøver oversat for dig til cifre: 1 og 0, og så er det pludselig let at skabe analogier mellem mikrobiomet og folks sundhed til havenes mikrobiom og planetens sundhed. Så vi stillede spørgsmålet: Tænk, hvis vi kunne studere planetens mikrobiom på samme måde som, vi studerer det menneskelige mikrobiom?"
I den model fungerer mikroorganismer som tidlige markører for forurening eller usunde økosystemer, der er analoge med kræftbiomarkører.
"Måske kan de på et eller andet tidspunkt bruges til at afhjælpe økosystemer, der er i dårlig form, svarende til sygdomsbehandling. Alt dette kræver forståelse af biodiversitet, og der er meget lidt kendt om mikroorganismerne i dag," uddyber Peer Bork.
Han besluttede sig derfor for at foretage en mikrobiel folketælling af vores planet. Han blev en del af Tara Oceans Consortium omkring en forskningssejlbåd, der samler plankton rundt om i verden på dybder ned til 1.000 meter. Herefter katalogiserer og analyserer han og hans kolleger de mikrobielle arter og deres gener i verdenshavene.
"Vi har nu skabt et mikrobielt referencekatalog for havene, der omfatter plankton, vira, bakterier og eukaryote organismer. Vi har også udviklet måder til at studere interaktioner mellem dem og til at kunne kvantificere, hvordan miljøfaktorer såsom temperatur påvirker havets mikrobielle samfund," fortæller Peer Bork.

Skonnerten Tara er blevet brugt til en række globale ekspeditioner inklusive dem i Arktis og Antarktis. Et stort forskningskonsortium (Tara Oceans) er forpligtet til at analysere de indsamlede prøver. Ekspeditionerne omfattede også træningsprogrammer og opsøgende aktiviteter for at øge offentlighedens bevidsthed om miljø- og klimabeskyttelse.
Værktøjerne skal være gode nok
Peer Bork tror på, at hans og kollegernes forskning kan skabe meget bedre evolutionær forståelse for dynamikken i de marine mikrobielle samfund, især i forhold til globale klimaændringer. Derfor har de indsamlet og delt alle de molekylære, morfologiske og miljømæssige data. Dette har virkelig udvidet deres forståelse af havets mikrobielle økologi.
"Vi kan nu forstå og vurdere de aktuelle ændringer i dette økosystem og forhåbentlig hjælpe med at genskabe det og derved hjælpe med at sikre vores planets fremtidige beboelighed. Vi analyserede også jordbunden rundt om i verden og opdagede en global krig mellem svampe og bakterier med antibiotika og antibiotikaresistensgener som henholdsvis våben og forsvarssystemer. Så vi kan sammenligne mikrobielle samfund i forskellige levesteder for at undersøge, hvordan individuelle gener og hele arter udvikler sig, og hvordan de spredes over hele kloden," forklarer Peer Bork.
Peer Borks rejse fra at arbejde med meget små proteindomæner, store genom-sekventeringsprojekter og hans studier af mennesket, havet og senest Jordens mikrobiom har gjort ham til en af de bedst kendte og hyppigst citerede forskere i verden. Faktisk har næsten alle, der analyserer genom- eller metagenomiske data, brugt en metode eller et værktøj udviklet eller påvirket af Peer Bork. Dette motiverer virkelig denne store videnskabelige værktøjsmager.
"Du gør altid en særlig indsats for at sikre, at andre faktisk kan bruge det, du udvikler. Værktøjerne skal være gode nok til at analysere data, men de skal også være intuitive og nemme at bruge. Hvis du har brugt tid på at skabe et værktøj og har delt det med andre, er det meget tilfredsstillende at vide, at andre mennesker bruger det og deler deres egne data, så vi kan hjælpe hinanden med at skabe en fremtid med en sundere global befolkning og mere bæredygtig planet," lyder det afslutningsvis fra Peer Bork.
